Autoria: Tomás Vieira (LEMec)
Com a recente oficialização dos novos laureados Nobel, o Diferencial procurou quem melhor entende do assunto dentro do Instituto Superior Técnico (IST). Em entrevista, Simão Rocha conta-nos a história dos microRNAs e a revolução que constituíram para a biologia molecular, e no fundo, para a genética molecular.
O dia 10 de dezembro é marcado todos os anos pela cerimónia de entrega, dos prémios Nobel, os quais reconhecem o trabalho realizado nas áreas da Física, Química, Medicina, Literatura, Economia e Paz. O Diferencial contactou diversos professores e investigadores no Técnico para comentar sobre o trabalho dos galardoados.
Simão Rocha, professor auxiliar do departamento de Bioengenharia e líder do Laboratório de Epigenética de Células Estaminais do Grupo de Engenharia de Células Estaminais (SCERG), inserido no Instituto de Bioengenharia e Biociência (iBB) do IST, revelou-se a escolha certa para abordar o tema.
Teoria e importância
No conhecido dogma central da biologia molecular [1] temos estabelecidas as relações e processos entre as três principais biomoléculas (DNA, RNA e proteínas) que ditam a vida.
As diferentes etapas de conversão nele descritas são sempre alvo de controlo para que, como pretendido, haja apenas expressão dos genes de interesse, naquela célula e naquele momento. Isto pois, pelo absurdo, o gene necessário à produção de lactase, a enzima protéica hidrolítica da lactose (açúcar presente no leite de materno e de vaca), não é nunca necessário nos nossos neurónios, por mais que estes, e todas as nossas células, possuam a informação necessária para a sua produção. Ainda neste conceito, o fator temporal é muito importante, uma vez que a expressão de um gene específico não tem de ser constitutiva, ou seja, durante todo o ciclo de vida de um ser vivo. Estes dois aspetos de forma muito generalista levantam o imperativo da regulação genética.
Outro aspecto subjacente ao dogma, advindo do facto de se tratar de uma simplificação, é que os principais atores moleculares têm um papel muito fixo. No caso do RNA é o transporte e mediação do fluxo de informação genética do DNA para a proteína correspondente. No entanto, as descobertas feitas ainda nos anos 90 por Victor Ambros e Gary Ruvkun revelaram uma nova faceta desta biomolécula, adjacente a um novo subtipo chamado microRNAs. [2] Isto salvaguardando o caso dos snRNAs, descobertos na década de 60 e responsáveis pela formação do spliceossoma [3] cuja função não se enquadra nos moldes mais genéricos.
“Já eram conhecidos os RNAs clássicos: os ribossomais, mensageiros e os de transferência, mas começou-se a perceber que havia muito mais RNAs reguladores”, comenta Simão Rocha.
Resumidamente, estes novos RNAs são sequências muito curtas (com uma média de 22 ribonucleotídeos [4]) que se ligam, por complementaridade, a uma sequência de RNA recém traduzida. A interação por se dar exatamente por complementaridade, ou seja, correspondência base a base entre as duas cadeias, tem a ela associada um elevado grau de especificidade. Nisto, e após a conjugação dos dois RNAs (mRNA e microRNA), dão-se dois diferentes destinos para o complexo formado: sinalização para a sua degradação ou inibição direta da tradução do mRNA a que se ligou o microRNA. Genericamente, o que resulta da ação desta “nova” biomolecula é a não expressão de um gene numa proteína.
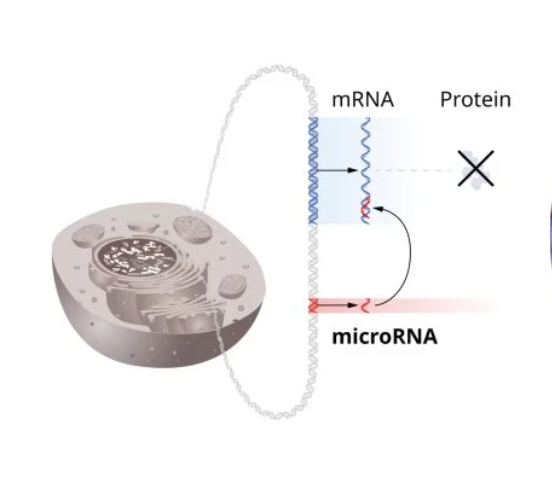
“O que se percebeu com esta descoberta dos microRNAs é que havia aqui um nível de regulação que era não tanto na primeira parte: do DNA para o RNA [transcrição], no fundo era entre o RNA e a proteína”, finalizando a explicação com: “Do ponto de vista de biologia básica, são RNAs que atuam num outro nível de regulação genética.”
A intervenção destas biomoléculas neste patamar genético confere-lhes o título de agentes na regulação pós-transcricional, uma vez que o seu mecanismo de ação é de silenciamento de mRNAs. Globalmente, a este tipo de RNA que silenciam o RNA mensageiro chamamos, RNA de interferência (RNAi), grupo que também inclui os small interfering RNAs (siRNA) posteriormente descobertos [5]. Estes últimos apresentam um mecanismo afim, mas distinto e reforçam a ideia pintada pelo investigador: “Já se sabia que havia regulação a vários níveis, mas esta era completamente diferente”.
Diferenciação celular e células estaminais
Um dos axiomas biológicos e aspetos já referidos é a presença da mesma informação genética, virtualmente, em todas as nossas células somáticas. Isto, juntamente com o conceito de totipotência celular, fez incidir alguma luz sobre um dos campos mais complexos da biologia. Fundamentalmente, “os microRNAs são muito importantes não só para a diferenciação de células estaminais como também para o próprio desenvolvimento embrionário”. No entanto, a própria expressão destes é regulada e difere consoante o tecido e estágio de desenvolvimento: “Há classes de microRNAs que estão mais expressas no desenvolvimento embrionário e depois, outras estão mais expressas nas células já diferenciadas”, esclarece Simão Rocha. Portanto, há uma regulação da regulação genética, muito assente no facto óbvio que estes RNAs têm de ser transcritos. “O processamento dos microRNAs também já está muito estudado e é muito conhecido”, ao que o investigador acrescenta a curiosidade: “Há muitos destes microRNAs que não estão isolados no nosso genoma, são de regiões em que se produz um grande pré-microRNA que depois é processado em vários microRNAs diferentes.”
“Nós sabemos que há uma flutuação de diferentes classes de microRNAs durante o desenvolvimento”, ao que Simão Rocha completa dizendo que este aspecto se transpõe para a investigação dado que knockouts ou outros silenciamentos alterarão padrões de desenvolvimento e, futuramente, servirão como ponte para o esclarecimento de enigmas nesta área. Com este conhecimento, abre também a hipótese de uma engenharia celular mais direta: “Poderemos vir a guiar uma célula pluripotente a se diferenciar num tipo celular especializado guiado por microRNAs, entre outros fatores”. Ao que, pertinentemente, completa, esclarecendo que a possibilidade de se prosseguir no sentido inverso é uma realidade: “há vários protocolos, chamados protocolos de reprogramação, que consistem em tornar uma célula somática numa célula estaminal, estes podem agora ser acelarados expondo a célula a um cocktail de microRNAs”
Direcionando o foco para a patologia, foi ainda falado brevemente da relação destas moléculas para com o cancro: “Sabe-se, hoje em dia, que há biomarcadores de certos cancros que são microRNAs que estão sub ou sob-expressos”.
Junk DNA e história
Ainda que esta classe nunca configure um gene na sua definição mais clássica (codificante para proteína), o papel biológico que desempenha fá-lo ser verdadeiramente um gene funcional – gene no sentido lato. Este aspeto é particularmente pertinente quando olhamos para a história da sequenciação do genoma humano, isto pois, durante muitos anos, o DNA sem função conhecida era apelidado de junk DNA. O termo quase pejorativo tem vindo a ser reconhecido como um reflexo da ignorância e, mais uma vez, a descoberta aqui falada está ativamente a desconstruir essa mesma ideia.
Contudo, para Simão Rocha, “o mistério não está completamente resolvido” mas “a noção do junk DNA é muito diferente”, completando com o exemplo dos transposões, sequências por vezes não codificantes e por vezes de origens víricas, apelidadas anteriormente de inúteis. Finaliza referindo: “nós já não achamos que é só lixo, até porque seria um desperdício que a maior parte do nosso genoma, cerca de 98%, fosse quase só lixo, acha-se que há nele função. O problema é perceber essa função”.
Acrescenta ainda o esclarecimento para a biologia que a conquista constituiu: “Sem se saber da existência de microRNAs, pensava-se que não era um mecanismo muito usual, mas na verdade o RNA de interferência é algo que as células usam e abusam.” Isto porque, um dos conhecimentos mais importantes extraídos da descoberta foi a universalidade e alta conservação do mecanismo. Em outros termos, nada do falado é biologicamente novo, existe e foi essencial para a atual complexidade biológica.
Certo… mas e no Técnico?
Por mais que o professor não conseguisse assegurar a existência de atuais investigações a decorrer dentro do IST envolvendo diretamente microRNAs, facultou-nos que dentro do iBB trabalhou com RNAs de interferência. A ideia geral revelou ser análoga, uma vez que a expressão ectópica de RNAs antisense permite diminuir a expressão, técnica utilizada pelo investigador para estudar um fator que se liga e regula a estrutura da cromatina [6]
Apesar disso, o investigador discursa sobre os seus planos para um futuro projeto envolvendo já os conhecimentos deste prémio. A investigação, se tudo correr bem, incidirá sobre uma doença do foro do neurodesenvolvimento, causada pela deficiente expressão/inexistência de um gene (UBE3A) regulado por imprinting parental, expresso apenas na cópia materna. A doença chamada síndrome de Angelman é causada pela perda da cópia materna do UBE3A, contudo a cópia paterna do gene UBE3A continua presente ainda que não seja expresso. Oportunamente para a construção de uma cura, este silenciamento é causado pela expressão de um RNA antisense ao do mRNA do gene, silenciando-o. Nisto, se os investigadores conseguirem interferir com o RNA antisense produzido naturalmente permitirá a reativação da cópia do gene paterno, única nestes indivíduos. Em suma, a ideia geral é restituir a expressão do gene usando microRNAs que terão como alvo o RNA antisense, restituindo assim a expressão do UBE3A nos neurónios dos indivíduos com esta doença.
Notas finais
Com a explicação e contexto histórico articulados, o professor remata a entrevista com “o reconhecimento é super merecido”. Todavia, enfatiza o seu atraso: “chegou agora, em 2024, demorou mais de 20 anos, mas é normal: entre uma grande descoberta e até se ganhar o prémio Nobel passa muito tempo.” Em contrapartida, refere um exemplo recente, também biológico, mas vencedor do prémio Nobel da Química: “Há casos que foi muito mais rápido, um deles é o caso da tecnologia do CRISPR-Cas9”, uma vez que esta descoberta data 2012 e a atribuição do Nobel foi no ano de 2020. Atribui este desfasamento à demora na aplicação, quando comparado com o exemplo anterior, cujo qual já o principal e mais potente motor de edição genética. Como nota final, destaca as numerosas investigações, ainda prematuras, a serem feitas atualmente com esta descoberta, na clínica. Tal somado aos planos dentro iBB neste sentido ilustram um futuro cheio de microRNAs e onde a exploração deste mecanismo trará resposta a diversos desafios da medicina.
Stem Cell Epigenetics Lab
(https://scerg.tecnico.ulisboa.pt/biolengpsclab3.html)
Stem Cell Engineering Research Group (https://ibb.tecnico.ulisboa.pt/research/stem-cell-engineering-scerg/)
Institute for Bioengineering and Biosciences (https://ibb.tecnico.ulisboa.pt/)
[1] – Elaine A. Ostrander. (2024, December 9). Central Dogma. National Human Genome Research Institute. https://www.genome.gov/genetics-glossary/Central-Dogma
[2] – Nobel Prize Outreach. (2024, December 9). Press release. The Nobel Prize. https://www.nobelprize.org/prizes/medicine/2024/press-release/
[3] – Nucleic Acids and Molecular Genetics: Human Biochemistry (Second Edition, chapter 10). Gerald Litwack. (2022) https://doi.org/10.1016/B978-0-323-85718-5.00010-8.
[4] – O’Brien, J., Hayder, H., Zayed, Y., & Peng, C. (2018). Overview of MicroRNA Biogenesis, Mechanisms of Actions, and Circulation. Frontiers in endocrinology, 9, 402. https://doi.org/10.3389/fendo.2018.00402
[5] – Dana, H., Chalbatani, G. M., Mahmoodzadeh, H., Karimloo, R., Rezaiean, O., Moradzadeh, A., Mehmandoost, N., Moazzen, F., Mazraeh, A., Marmari, V., Ebrahimi, M., Rashno, M. M., Abadi, S. J., & Gharagouzlo, E. (2017). Molecular Mechanisms and Biological Functions of siRNA. International journal of biomedical science : IJBS, 13(2), 48–57. https://pmc.ncbi.nlm.nih.gov/articles/PMC5542916/
[6] – Jarid2 Is Implicated in the Initial Xist-Induced Targeting of PRC2 to the Inactive X Chromosome.da Rocha ST, Boeva V, Escamilla-Del-Arenal M, Ancelin K, Granier C, Matias NR, Sanulli S, Chow J, Schulz E, Picard C, Kaneko S, Helin K, Reinberg D, Stewart AF, Wutz A, Margueron R, Heard E.Mol Cell. 2014 Jan 23;53(2):301-16. doi: 10.1016/j.molcel.2014.01.002.PMID: 24462204 Free




